Rispondo qui alla domanda fatta da Riccardo in un'altra discussione:
ricki51 ha scritto:
Approfitto di questo dialogo per chiederti se conosci qualche ragione per cui le ricerche genetiche sulle Epipactis, al contrario di altri generi, sembrino incontrare grosse difficoltà. Noi abbiamo collaborato per molti anni con il prof. Cozzolino, per fornire materiale di sicura identificazione su cui effettuare le analisi. Ma pare che al momento gli studi abbiano ancora bisogno di tempo.
Te lo dico perché a me è venuto un dubbio: e se Epipactis fosse un genere di una singola specie?
Orco che scoop: atrorubens, palustris e nordeniorum subsp. maricae tutte variabilità di helleborine!
Questo spiegherebbe bene gli imbarazzi dei genetisti e offrirebbe pure una soddisfazione irripetibile a parecchi amici del Giros. Solo ti chiedo, nel caso nemmeno tanto assurdo che la genetica possa arrivare a una conclusione del genere, noi saremmo ancora autorizzati a catalogare le differenze fenotipiche, oppure verremmo relegati in un limbo di inutilità e disperazione?
Pondera bene la tua risposta, perché non rispondo delle reazioni di Luciano, quando gli comunicherò la cosa. Ciao Riccardo
Come dicevo, facendo una rapida ricerca tra le pubblicazioni recenti, ho trovato un lavoro proprio di Cozzolino. Non so dire con certezza se tale lavoro rappresenti la pubblicazione dei risultati ottenuti, anche dalla vostra collaborazione (sono riportate varie località anche dell'Emilia Romagna, per cui potrebbe essere), ma direi che per la complessità delle indagini svolte, è sicuramente un lavoro interessante, punto di partenza, per noi, per fare alcune considerazioni sulle Epipactis di casa nostra!
Innanzitutto l'articolo, che è scaricabile online. Eravate a conoscenza di questa pubblicazione?
V. Tranchida-Lombardo, D. Cafasso, A. Cristaudo and S. Cozzolino, (2010) - Phylogeographic patterns, genetic affinities and morphological differentiation between Epipactis helleborine and related lineages in a Mediterranean glacial refugium. Annals of Botany, Page 1 of 10.Supplemental Table -
Supplemental Figure.
Dopo una rapida lettura, lo confesso un po' superficiale, appare chiaro che gli autori sono andati ad analizzare geneticamente molte specie di Epipactis, provenienti da molteplici stazioni e località, italiane ed estere, e ne abbiano identificato aplotipo e ribotipo, basandosi sulle sequenze del DNA plastidico e del RNA ribosomale. Qui ci sarebbe da aprire una ulteriore, lunga, discussione per spiegare di cosa stiamo parlando, e perchè la scelta è ricaduta proprio su queste sequenze, ma dovremmo affrontare temi piuttosto complessi e specialistici. Per cui, in modo più semplice e sintetico possibile, possiamo dire che:
I
plastidi sono particolari organuli della cellula vegetale. All'interno dei plastidi è presente una matrice (stroma) cioè un compartimento idrofilo contenente DNA, ribosomi, precursori metabolici, prodotti finali ed enzimi abilitati al funzionamento biochimico dell’intero organulo. Il
DNA plastidiale è un filamento circolare di 0,04mm-0,3 mm di lunghezza caratterizzato da una marcata presenza della coppia guanosina-citosina. Avendo un DNA proprio e dei propri ribosomi i plastidi sono degli organuli semi-autonomi in quanto possono sintetizzare delle proteine indipendentemente dall'informazione genetica del genoma nucleare. Il DNA plastidico è ereditato per via materna.
L'
aplotipo è la combinazione lungo un cromosoma, segmento cromosomico, o filamento di DNA, di una sequenza di geni strettamente e statisticamente associati tra di loro (linkage disequilibrium), e che in genere, vengono ereditati insieme, costituendo di solito una particolare sequenza (aplotipo) ancestrale, diffusa nella popolazione in cui è rilevato, perché trasmesso lungo la discendenza da un comune progenitore. In questo caso è stato analizzato l'aplotipo del DNA presente all'interno dei plastidi, ovvero il DNA plastidico o plastidiale.
Il
ribotipo invece, analogamente al genotipo che è il contenuto della informazione genica scritta in DNA, è il contenuto della informazione genica, ma espressa in RNA. Il DNA infatti è solo uno dei due acidi nucleici contenuti nella cellula, l'altro è l'RNA. Il DNA prima di essere tradotto in proteine, viene trascritto in RNA messaggero (mRNA), una molecola intermedia, che consente alla cellula di leggere cosa c'è scritto nel DNA. Ad un singolo filamento di DNA spesso possono corrispondere svariati filamenti di RNA (ribotipi), nel senso che la cellula può leggere e trascrivere lo stesso filamento di DNA in vari modi diversi! Esistono poi l'
RNA ribosomale (rRNA) (RNA che va a costituire i ribosomi, ovvero particolari strutture altamente conservate, e deputate a tradurre l'RNA messaggero in proteine, ed è a questo tipo che di RNA che si fa riferimento in questo studio) e infine l'RNA transfer (t-RNA) che trasporta gli amminoacidi (unità strutturale di base delle proteine) ai ribosomi. Esistono anche alcuni altri tipi di RNA negli eucarioti ma possiamo tranquillamente trascurarli!
Venendo all'articolo, o meglio a quello che più ci può interessare di esso, facendo varie analisi gli autori hanno individuato per ogni specie, come dicevamo, vari aplotipi e ribotipi, riassunti nella seguente tabella. Gli aplotipi sono stati identificati con la lettera E (E1-E19) mentre i ribotipi con la lettera R (R1-R7).
Allegato:
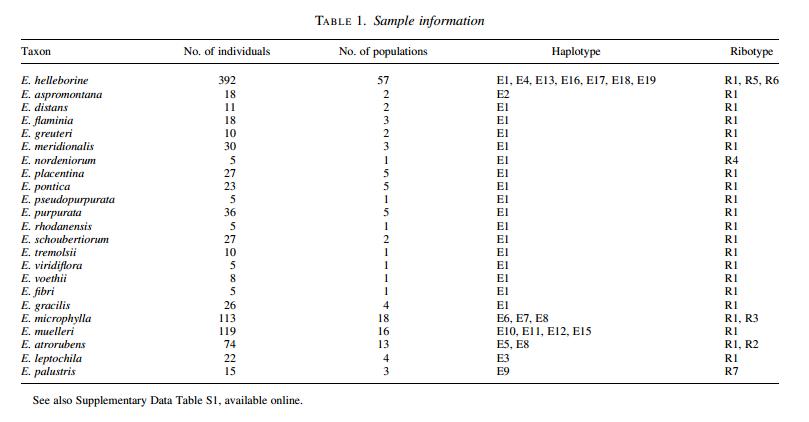 aplotipi e ribotipi.JPG [ 39.01 KiB | Osservato 14840 volte ]
aplotipi e ribotipi.JPG [ 39.01 KiB | Osservato 14840 volte ]
Nell'articolo viene presentata un'altra tabella in cui sono rappresentate le differenze di sequenza tra i vari aplotipi identificati, ovvero le mutazioni significative rilevate, ma probabilmente molto più interessanti, sono le tabelle subito successive, con la rappresentazione dei dati ottenuti dalle analisi.
Allegato:
 mutazioni.JPG [ 54.67 KiB | Osservato 14828 volte ]
mutazioni.JPG [ 54.67 KiB | Osservato 14828 volte ]
Albero filogenetico basato sui ribotipi. (Ho aggiunto direttamente i nomi delle specie a cui la sigla del ribotipo o aplotipo fanno riferimento).
Allegato:
 tree.JPG [ 35.22 KiB | Osservato 14840 volte ]
tree.JPG [ 35.22 KiB | Osservato 14840 volte ]
Network d'analisi basato sugli aplotipi.
Allegato:
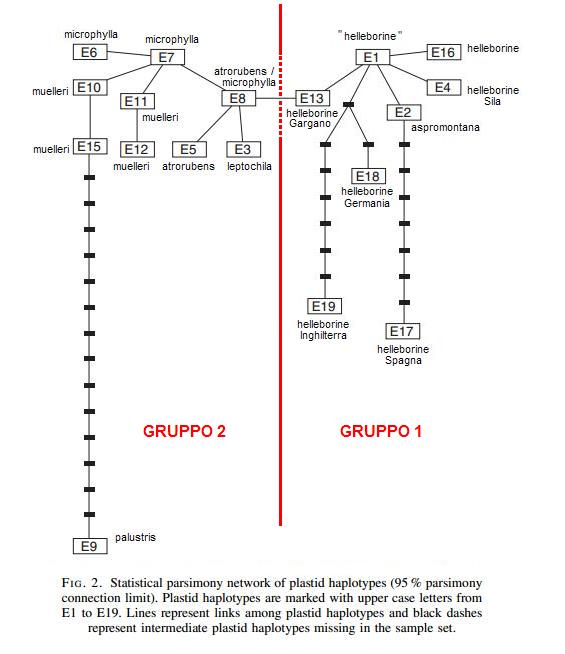 tree2.JPG [ 42.39 KiB | Osservato 14834 volte ]
tree2.JPG [ 42.39 KiB | Osservato 14834 volte ]
Discussione e risultati. (
traduzione dell'articolo di Cozzolino et al., più alcune considerazioni personali)
Gli autori non parlano di Epipactis come di
un genere di una singola specie, ne tanto meno di atrorubens, palustris e nordeniorum subsp. maricae tutte variabilità di helleborine, bensì dimostrano di aver individuato sulle popolazioni studiate una serie di aplotipi e ribotipi che consentono di avanzare delle considerazioni interessanti e delle ipotesi sulla evoluzione filogeografica del genere Epipactis, che sembra essere piuttosto complessa!
Il primo risultato da non sottovalutare è che la sequenza utilizza di DNA plastidico, rbcL–accD intergenic spacer, è stata utile per fare considerazioni ed analisi di tipo filogeografico, ma poco utile per avanzare ipotesi di filogenesi delle specie, dato che è stata trovata un'unica mutazione a singola base (in posizione 259 nel allineamento dell'aplotipo plastidico) che consente però di individuare due principali gruppi di specie (il gruppo di E. helleborine da una parte ed un secondo gruppo formato da E. atrorubens /E. microphylla / E. muelleri / E. leptochila).
In questo caso, tenuto conto della ampia distribuzione di queste specie e della presenza di popolazioni talvolta anche piuttosto numerose, è possibile ipotizzare che la bassa variabilità del DNA plastidico, almeno in termini di sostituzioni di basi, è probabilmente dovuto al differenziamento recente delle varie specie, piuttosto che ad un "basso tasso di mutazione" o a fenomeni di deriva genetica, quali ricorrenti "colli di bottiglia", fenomeni che fortemente riducono la variabilità dell'aplotipo plastidico.
[Inciso: Il fenomeno del "collo di bottiglia" nella
deriva genetica, è un fenomeno che si verifica quando il numero di individui facenti parte di una popolazione, viene ridotto drasticamente da forze atipiche nella selezione naturale (distruzione della quasi totalità di una popolazione per eventi esterni) o ne viene isolata definitivamente una parte, (spostamenti migratori anomali, barriere geografiche). Ovviamente se solo una parte esigua della popolazione generale sopravvive, o comunque sopravvive isolata dal resto della popolazione, tali sopravvissuti potranno, per il passaggio attraverso un "collo di bottiglia" metaforico dal quale risultano unici o isolati, rigenerare la popolazione originale avendo a disposizione solo il proprio corredo genetico, che non può essere significativo di tutta la popolazione generale della propria specie di origine, anzi sarà molto limitato. La conseguente riduzione della variabilità genetica può giungere alla criticità e tendere ad eliminare del tutto alcuni alleli (caratteri fenotipici), ma anche a far sì che altri vengano rappresentati in eccesso nel pool genico. Alcuni caratteri fenotipici della specie potranno dunque scomparire o altri essere espressi in modo eccessivo.
La deriva genetica ha come effetto quello di far divenire un allele e dunque il fenotipo da esso rappresentato, più comune o più raro col passare di generazioni successive. Pensando alla divergenza o alla presenza/assenza di certe gibbe, o al campo basale diviso o indiviso o a forma di ali di farfalla, o alla cavità stigmatica più o meno ampia, mi viene da pensare che certe popolazioni di Ophrys fuciflora che "ci" appaiono tanto differenti le une dalle altre, morfologicamente, non siano poi così diverse!

]
Tornando alle Epipactis, il modello filogeografico di questo studio, mostra un singolo aplotipo plastidiale principale per ciascuna specie, caratterizzata da un'ampia distribuzione geografica nella penisola italiana: E. helleborine (E1), E. muelleri (E10), E. microphylla (E7), E. leptochila (E3), E. atrorubens (E5), e alcuni aplotipi unici o rari con distribuzioni localizzate. I cinque aplotipi plastidici ampiamente diffusi possono essere suddivisi in un network di analisi in due gruppi principali: Uno composto dai quattro aplotipi plastidici (E10, E7, E3 ed E5) e l'altro composto solamente da E. helleborine (s.s. e s.l.) con aplotipo plastidiale E1 .
Risulta dunque che, volenti o nolenti, E. muelleri, E. microphylla, E. leptochila ed E. atrorubens, sono tutte strettamente correlate (uno o due step mutazionali), e risultano anche vicine ad altri aplotipi plastidici poco diffusi (E8) condiviso da certe E. microphylla e E. atrorubens, o geograficamente localizzati (E11, E12, E15 ed E6, ovvero in ordine E. muelleri i primi tre ed E. microphylla E6). Epipactis microphylla e E. atrorubens possono condividere un aplotipo plastidico comune (E8) fatto che non può essere facilmente spiegato come conseguenza del flusso genico tra le due specie, che possiedono differenti ribotipi nucleari. Una potenziale alternativa per spiegare questo modello è che l'aplotipo plastidiale E8 rappresenta l'aplotipo plastidico ancestrale di queste due specie di recente evoluzione. Questa ipotesi è supportata dal network di analisi, in cui si osserva la posizione intermedia di E8 tra gli aplotipi plastidici E5 (tipico di E. atrorubens) e E7 (tipico di E. microphylla) e dal fatto che gli individui di E. microphylla ed E. atrorubens con aplotipo plastidiale E8 hanno sempre il ribotipo ancestrale R1.
Una alternativa per spiegare questo accoppiamento non selettivo, tra genomi nucleari e citoplasmatici, sarebbe quella di ipotizzare la presenza di incroci e autofecondazione tra popolazioni di queste due specie, allogame facoltative.
Le strette connessioni tra gli aplotipi presenti in Sardegna e quelli dell'Italia centrale può essere attribuita a cicli glaciali del Quaternario, che, a causa dei presunti collegamenti che a quel tempo erano presenti tra il blocco Corso-Sardo e l'attuale Toscana, hanno permesso uno scambio biotico attraverso l'arcipelago toscano. L'attuale diversità di aplotipi plastidiali in Sardegna è rappresentato da una combinazione di due aplotipi unici (E11 ed E15, di E. muelleri) e aplotipi condivisi con la terraferma (E1, E8, ed E10). Questi ultimi casi riguardanti gli aplotipi E8-E10 ad ampia diffusone(di E. muelleri, E.microphylla ed E.atrorubens della pensinsula italiana) ed E1 (caratteristica di E. helleborine sempre della Italia peninsulare). Inoltre, i due aplotipi sardi geograficamente localizzati (E11 e E15) sono strettamente collegati all'aplotipo E7 della terraferma, ampiamente diffuso, e al raro aplotipo E12 (una singola mutazione) presente solo in Toscana e nelle isole dell'Arcipelago Toscano (Isola d'Elba), che presumibilmente ha agito da ponte tra l?italia centrale e la Sardegna.
La E. helleborine s.s. è caratterizzata da un unico aplotipo plastidiale ampiamente diffuso (E1) distribuito in tutta la penisola italiana, dalle Alpi alla Sicilia. Strettamente legato a questo aplotipo plastidiale principale (solo un passo mutazionale) ci sono alcuni aplotipi plastidici (E13, E2, E4 ed E16) con una distribuzione geograficamente localizzata (E4 è presente solo nelle montagne della Sila e E13 solo sul promontorio del Gargano).
Sorprendentemente, aplotipo plastidiale (E1) di E. helleborine ampiamente diffuso in Italia, ha mostrato molte differenze rispetto agli aplotidi plastidiali della E. helleborine s.s. prelevati sia in Germania settentrionale e meridionale (E18, tre mutazioni), Inghilterra (E19, otto mutazioni) e Spagna (E17, nove mutazioni).
Le differenze (dieci mutazioni) tra gli aplotipi di E. helleborine provenienti da Italia, Inghilterra e Spagna, confermando la presenza di un segnale filogeografico significativo in questa regione del plastidio, suggeriscono anche che esse rappresentano tutte differenti rotte di colonizzazione che non si sono più incontrate dopo l'ultima espansione post-glaciale.
Il minor numero di differenze tra gli aplotipi delle E. helleborine italiane e tedesche indica probabilmente che i due ceppi che hanno colonizzato l'Europa centrale e l'Italia, sono partiti da un antenato comune. Queste evidenze filogeografiche indicano che in Italia sono presenti solo due ceppi principali, uno composto da E. helleborine s.s. ed E. helleborine s.l. (per lo più caratterizzate dall'aplotipo plastidiale E1) e un secondo ceppo che comprende E. muelleri, E. microphylla, E. atrorubens ed E. leptochila.
Nel primo gruppo, imparentate strette di
E. helleborine troviamo dunque:
E. aspromontana (E helleborine subsp. aspromontana); E. distans (E. helleborine sub. orbicularis); E. flaminia, E. greuteri, E. meridionalis, E. placentina,
E. pontica, E. purpurata, E. rhodanensis (E. bugacensis subsp. rhodanensis), E. schubertiorum (Epipactis helleborine subsp. schubertiorum), E. tremolsii (E helleborine subsp. tremolsii), E. voethii (E. helleborine subsp. helleborine), E. gracilis (E. exilis)
L'articolo prosegue con varie altre domande che gli autori si pongono ed ulteriori osservazioni, ma per ora mi fermo qui!

Allegato:
 mappa.JPG [ 82.42 KiB | Osservato 14839 volte ]
mappa.JPG [ 82.42 KiB | Osservato 14839 volte ]





